基于机器学习识别视网膜母细胞瘤中的关键基因及免疫浸润状况分析
关键词
摘要
目的:应用生物信息学方法探究视网膜母细胞瘤(retinoblastoma, RB)中的关键差异基因并进行富集分析、计算免疫浸润情况和预测敏感药物。方法:从基因表达综合数据库(gene expression omnibus, GEO)下载3个RB的基因表达芯片进行合并。使用R语言软件完成差异基因表达分析、基因富集分析、机器学习识别关键差异基因和免疫浸润分析。使用肿瘤免疫单细胞数据库 (tumor immune single-cell hub, TISCH)完成单细胞测序分析,应用DSigDB数据集进行药物富集分析。结果:肿瘤样本和正常样本间共有556个差异基因,基因本体论/京都基因与基因组百科全书(gene ontology/ kyoto encyclopedia of genes and genomes, GO/KEGG)富集显示这些共同基因参与了p53信号通路和细胞衰老等生物过程。 加权基因共表达网络分析(weighted gene co-network analysis, WGCNA)和支持向量机-递归特征消除(support vector machine - recursive feature elimination, SVM-RFE)机器学习联合分析得到12个关键特征基因。其中人类婆罗双树样基因1(spalt-like transcription factor 1, SALL1)在肿瘤样本中高表达,锌指蛋白385B(zinc finger protein 385B, ZNF385B)基因低表达。肿瘤免疫浸润分析揭示在RB中M1型巨噬细胞和自然杀伤细胞(natural killer cell, NK)细胞高于正常组织。单细胞分析验证了关键特征基因在不同细胞亚型中的表达差异,药物富集分析预测了格列本脲、环磷酰胺、硫唑嘌呤和大豆苷元等为潜在的敏感药物。结论:12个关键特征基因的异常表达与RB存在密切关联,这些关键特征基因可能对肿瘤免疫微环境存在重要影响,可作为潜在的治疗靶点。
全文
文章亮点
1 关键发现
• 本研究基于多组学分析发现了视网膜母细胞瘤12个关键特征基因,其中锌指蛋白家族基因[例如人类婆罗双树样基因1(spalt-like transcription factor 1, SALL1)基因和锌指蛋白385B(zinc finger protein 385B, ZNF385B)基因]与异常通路的活化,肿瘤免疫浸润等存在密切关联,进一步的探究显示格列本脲、环磷酰胺、硫唑嘌呤和大豆苷元可能是视网膜母细胞瘤潜在的敏感化合物。
2 已知与发现
• 视网膜母细胞瘤是儿童中最常见的眼内恶性肿瘤,RB1基因突变引起。视网膜母细胞瘤(retinoblastoma, RB)中的肿瘤细胞具有高度异质性,了解肿瘤内的异质性将有助于更好地了解该病的治疗反应和耐药性。基于现代多组学技术,我们可以通过基因和蛋白等层面了解RB中更复杂的分子机制,挖掘新的治疗靶点。
3 意义与改变
• 本研究揭示锌指蛋白家族基因可能是视网膜母细胞瘤潜在的新标志物(例如SALL1基因和ZNF385B基因),这些特征基因可作为潜在的研究靶点。格列本脲、环磷酰胺、硫唑嘌呤和大豆苷元等化合物可以作为潜在的敏感化合物进行后续的活性测试。
视网膜母细胞瘤(retinoblastoma, RB)是儿童中最常见的眼内恶性肿瘤,由位于人类染色体13q14.2区域的RB1基因突变引起[1-3]。在高收入国家,RB是一种高度可治愈的肿瘤,患者生存率超过99%,然而在许多低收入国家,大多数患者发生了致命性的肿瘤转移,预后不良[4-6]。RB的主要治疗目标首先是通过早期检测、治疗眼部肿瘤和预防转移扩散来挽救儿童的生命,次要目标是保护眼睛和保留视力。当前,RB的保守治疗方式已逐渐从全身放化疗转变为局部的眼动脉介入化学治疗(化疗)和玻璃体腔化疗,减少了很多药物不良反应,但是现有的治疗药物疗效仍然有限[4]。因此应用现代的多组学技术研究RB中新的有效的靶点,对于研发新的敏感药物具有重要意义。此外,RB中的肿瘤细胞具有高度异质性,了解肿瘤内的异质性将有助于更好地了解该病的治疗反应和耐药性[7-8]。现代的多组学分析技术,如高通量基因转录组测序、微阵列基因芯片测序、单细胞分析和蛋白组学等为深入探索RB的发病机制提供了更强有力的方法,为该病的诊断和治疗提供了重要的依据。本研究基于公共数据库,应用机器学习方法分析了RB中的关键特征基因并分析了重要的信号通路,在此基础上进一步探究了RB的肿瘤微环境并预测敏感药物,为RB的分子机制研究和新型药物开发提供了新的依据。
1 资料与方法
1.1 数据集获取
从基因表达综合数据库(gene expression omnibus, GEO) (https://www.ncbi.nlm.nih.gov/geo/) 下载GSE58780、GSE24673和GSE110811三个数据集,使用R语言(R-4.4.0, 64-bit)对数据集进行合并形成一个整合数据集,具体数据集信息详见表1。
|
GEO芯片编号 |
芯片类型 |
测序平台 |
样本量 |
|
GSE58780 |
微阵列芯片 |
GPL28718 |
59个RB样本,3个正常胎儿视网膜样本 |
|
GSE110811 |
微阵列芯片 |
GPL16686 |
28个RB样本,3个正常儿童视网膜样本 |
|
GSE24673 |
微阵列芯片 |
GPL6244 |
9个RB样本,2个正常儿童视网膜样本 |
1.2 方法
1.2.1 数据前处理
使用R语言中的limma(v 3.50.3)程序包对数据集进行基因表达量log2转换和取交集表达基因等标准化处理,并使用sva(v 3.42.0)程序包合并3个数据集,去除批次差异。
1.2.2 基因表达分析
使用limma程序包对RB肿瘤样本(n=96)和正常胎儿视网膜样本(n=8)间的差异表达基因(differentially expressed genes, DEGs)进行计算,DEGs的筛选标准为:logFC≥1 和校正P值< 0.05。
1.2.3 差异基因GO/KEGG富集分析
应用org.Hs.eg.db(v 3.14.0)、DOSE(v 3.20.1)和clusterProfiler(v 4.2.2)包对DEGs进行GO/KEGG分析。
1.2.4 WGCNA分析鉴定特征基因集
使用加权基因共表达网络分析(weighted gene co-network analysis, WGCNA) (v 1.73)程序包识别高度协同变化的基因模块,并探索这些模块与RB的关联,识别出关键的差异基因[9]。
1.2.5 支持向量机算法筛选关键差异基因
支持向量机(support vector machines, SVM)是一种二分类模型,适用于小样本、高维度的数据分类问题。使用e1071(v 1.7.13)程序包对差异基因进行支持向量分析,获取特征基因并进行可视化展示。pROC(v 1.18.5)程序包完成受试者工作特征曲线(receiver operating characteristic, ROC)分析。
1.2.6 免疫浸润分析
使用quanTIseq(v 1.4.1)程序包基于最小二乘法(lsei)的原理对10种免疫细胞亚型的表达矩阵进行反卷积计算,推测肿瘤组织中免疫细胞的构成情况。
1.2.7 GSVA分析
基因集变异分析(genesetvariationanalysis, GSVA)是一种非参数、无监督的算法,用于计算每个样本中特定基因集的富集得分并比较不同组间通路富集的差异。本研究应用GSVA方法分析与特征基因表达量密切相关的信号通路。
1.2.8 单细胞RNA数据分析
应用TISCH2(http://tisch.compbio.cn/home/)在线数据库分析RB(GSE166173)的单细胞测序数据,比较RB肿瘤样本和正常样本中特征基因的表达差异。TISCH2是一个专注于肿瘤微环境(tumor microenvironment, TME)的单细胞RNA测序数据库。该数据库提供单细胞水平的高精度细胞类型注释,支持跨不同癌症类型的肿瘤微环境深入探索[10]。
1.2.9 治疗药物富集分析
从药物特征数据库DSigDB(https://dsigdb.tanlab.org/DSigDBv1.0/)下载相关的药物-靶基因数据集,应用R软件分析RB特征基因与治疗药物存在的关联,构建基因集富集分析,预测潜在的治疗药物。DSigDB 基因集可与 GSEA 程序包相结合,用于将基因表达与药物联系起来,用于药物再利用和转化研究[11]。
1.2.10 统计学方法
所有统计分析均在R软件(R-4.2.0, 64-bit)中应用对应的程序包及默认的统计学方法进行计算。P<0.05表示差异具有统计学意义。
2 结果
2.1 差异基因分析
3个GEO数据集在合并前存在明显的批次差异,合并后批次差异得到有效的消除。基因表达分析显示,RB肿瘤样本和正常视网膜样本间存在556个差异基因。见图1。
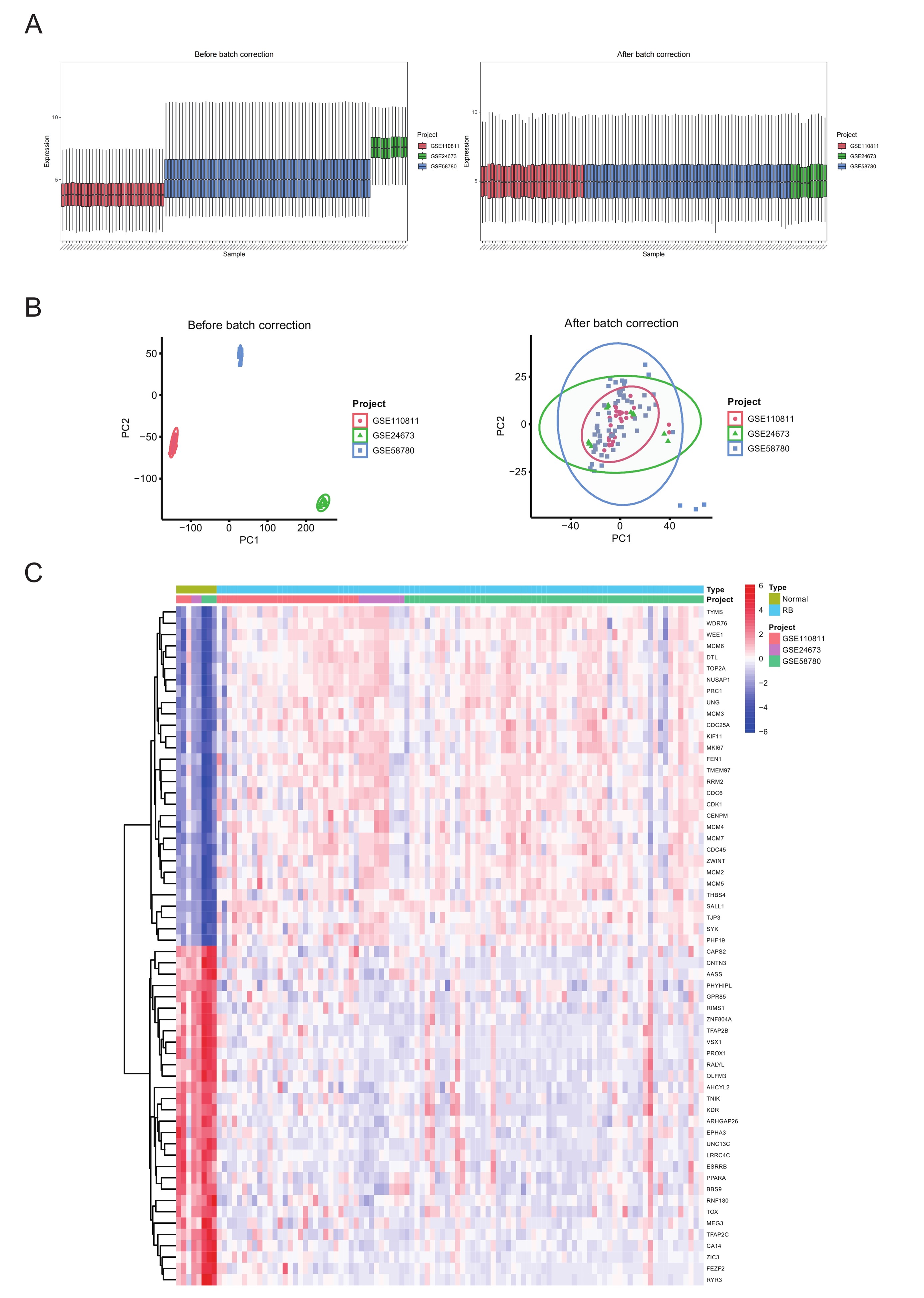
(A) 3个GEO数据集合并前后的箱线图;(B) 3个GEO数据集合并前后的PCA图,坐标轴代表的是每个样本标准化的主成分得分,反映样本在该主成分上的偏离程度;(C)基因表达差异热图(展示排名前30的差异基因),颜色代表每个基因的Z-SCORE,红色代表数值越高(高表达量),蓝色代表数值越低(低表达量)。
(A) Box plots of the three GEO datasets before and after batch effect correction. (B) Principal component analysis (PCA) plots of the three GEO datasets before and after integration. The axes represent standardized principal component scores for each sample, indicating their variance along the respective components. (C) Heatmap of the top 30 DEGs. The color scale represents Z-scores, with red indicating higher expression levels and blue indicating lower expression levels.
2.2 GO/KEGG基因富集分析
GO/KEGG分析预测556个差异基因参与的通路和生物学功能。见图2。
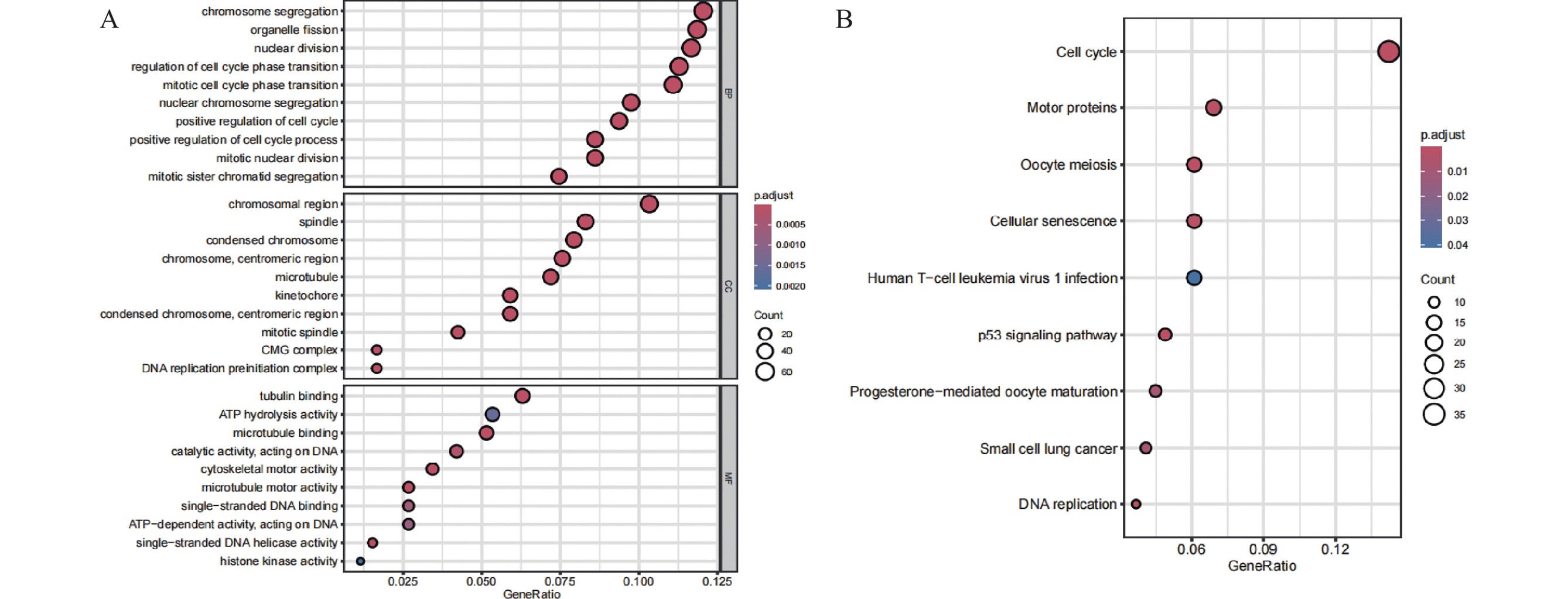
(A) GO基因功能富集分析;(B) KEGG基因功能富集分析。
(A) GO enrichment analysis. (B) KEGG pathway enrichment analysis.
2.3 WGCNA分析
应用WGCNA算法对数据集的所有基因进行分析,结果显示turquoise模块里的基因集与RB表型的相关性最高,共计853个特征基因。见图3。
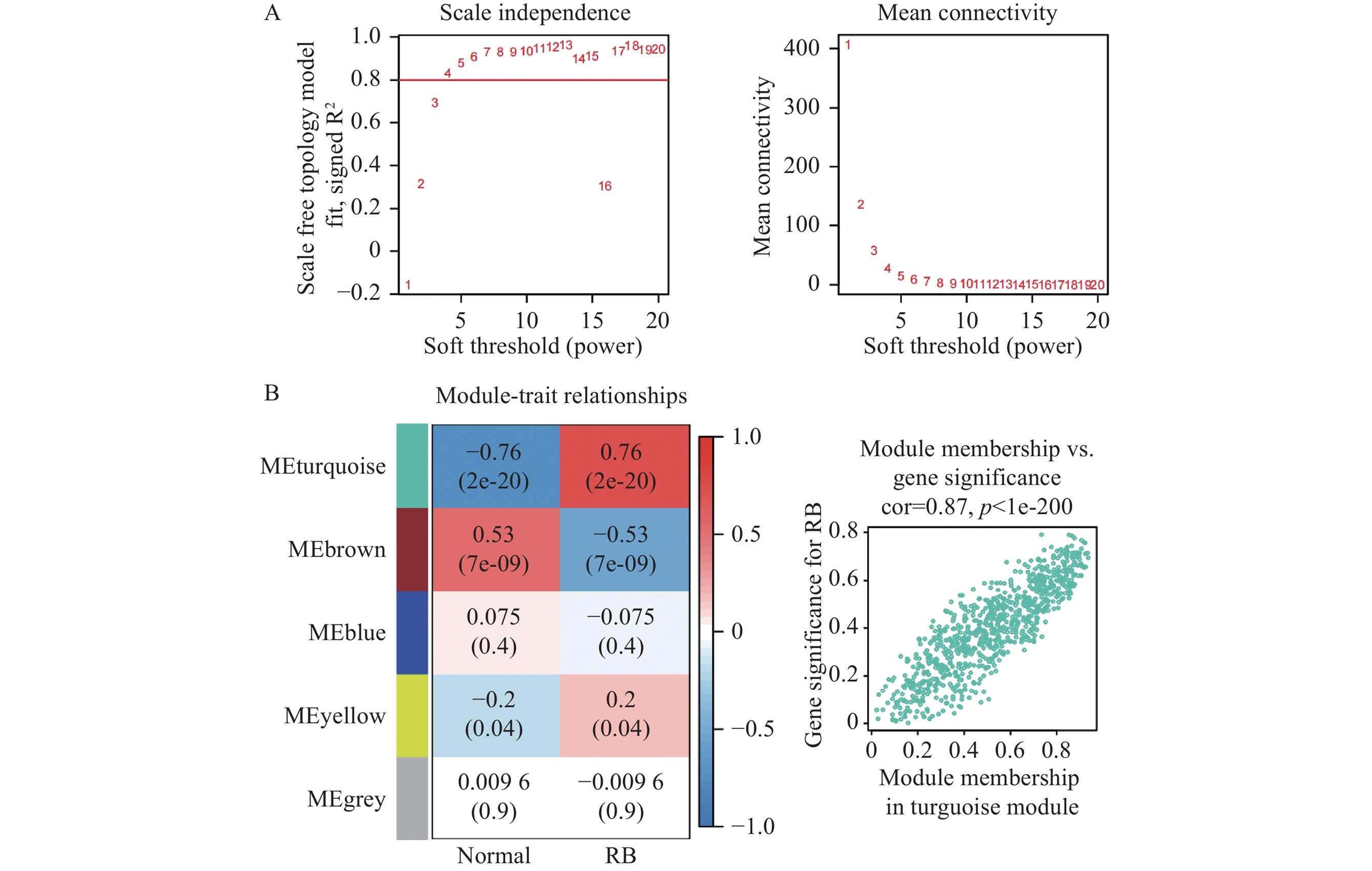
(A)尺度独立性和软阈值的平均连通性;(B)模块特征基因与临床表型相关性分析的热图。红色表示正相关,蓝色表示负相关。
(A) Scale-free topology fit index and mean connectivity analysis for various soft-thresholding powers. (B) Heatmap of module-trait associations. Red color indicates a positive correlation, and blue indicates a negative correlation.
2.4 SVM机器学习算法鉴定特征基因
在556个差异基因的基础上使用SVM机器学习算法计算出16个疾病特征基因(图4A),进一步与WGCNA特征基因取交集得到12个关键特征基因(图4B),这些关键特征基因在整合数据集中具有良好的诊断效能(图4C)。其中在RB肿瘤样本中上调的基因是人类婆罗双树样基因1(spalt-like transcription factor 1, SALL1)、二肽基肽酶4基因(dipeptidyl peptidase 4, DPP4)、细胞分裂周期蛋白20基因(cell division cycle 20, CDC20)、发状分裂增强子6基因(hairy and enhancer of split 6, HES6)、成蛋白2基因(formin 2, FMN2)、氯离子通道辅助蛋白1 (chloride intracellular channel 1, CLIC1)、热休克蛋白70家族A成员2基因[heat shock protein family A (Hsp70) member 2, HSPA2],下调的是胰岛素受体底物1基因(insulin receptor substrate 1, IRS1)、雌激素相关受体β基因(estrogen-related receptor β, ESRRB)、卷曲螺旋结构域包含蛋白146( coil-coil domain containing 146, CCDC146)、锌指蛋白385B (zinc finger protein 385B, ZNF385B)、膜联蛋白A4(annexin A4, ANXA4)(图4D-E)。
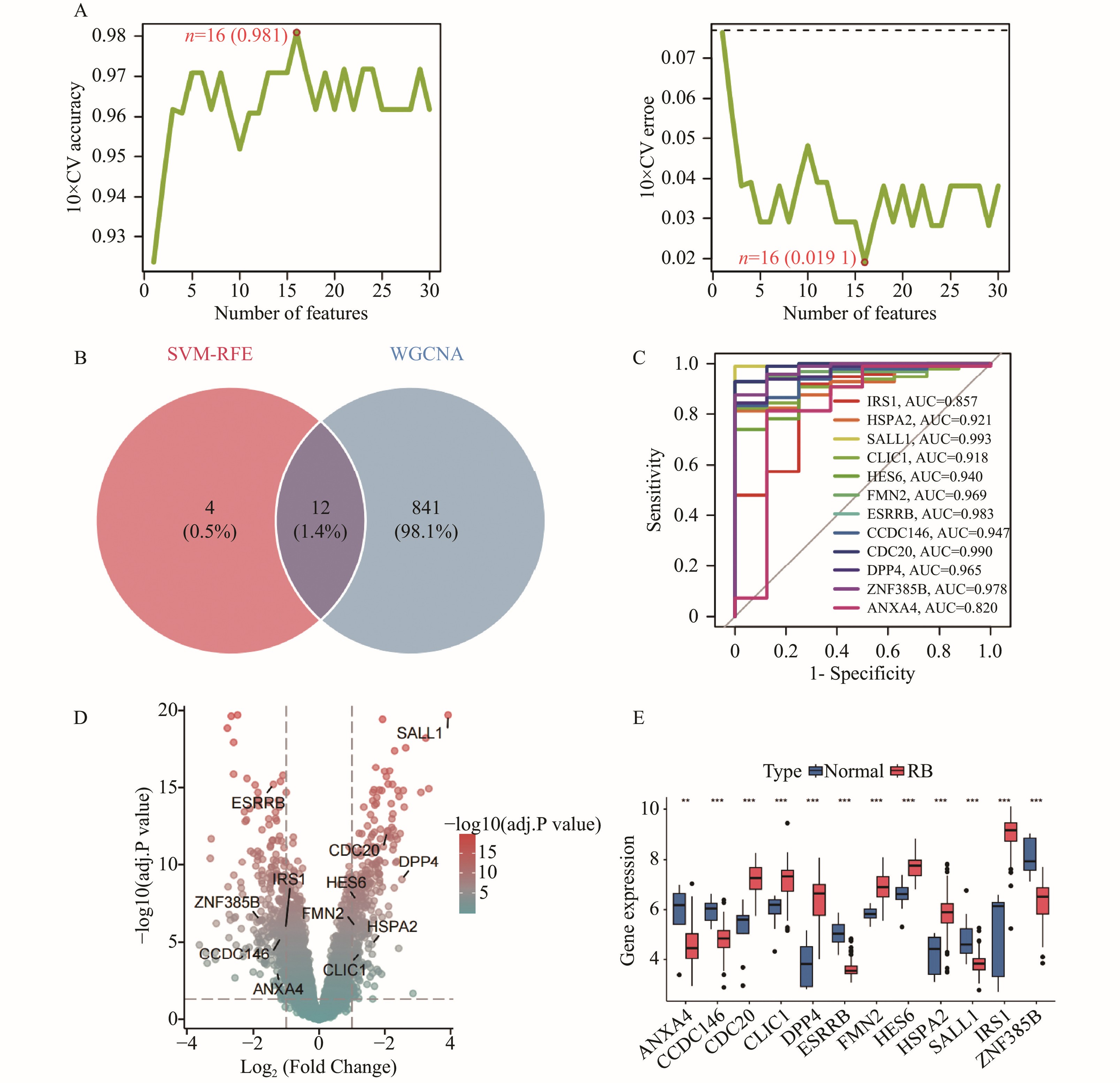
(A) SVM-RFE机器学习方法鉴定了16个疾病特征基因;(B)韦恩图显示SVM-RFE和WGCNA方法交集得到了12个关键疾病特征基因;(C) ROC曲线显示12个关键疾病特征基因在整合数据集中有良好的诊断效能;(D)火山图揭示12个关键特征基因的表达趋势;(E)箱式图揭示12个关键特征基因在肿瘤和正常样本中的表达差异。
(A) The SVM-RFE algorithm identified 16 disease feature genes. (B) A Venn diagram shows 12 key disease feature genes at the intersection of the SVM-RFE and WGCNA methods. (C) The ROC curve demonstrates the robust diagnostic performance of the 12 key feature genes in the integrated dataset. (D) A volcano plot reveals the expression trends (up- or down-regulation) of the 12 key feature genes. (E) Box plots illustrate the differential expression of the 12 key feature genes between tumor and normal samples.
2.5 GSVA分析
由前面的分析可知12个关键特征基因中肿瘤样本SALL1的上升趋势最明显,ZNF385B下降趋势最强,在此基础上,应用GSVA分析寻找与这两个基因关联性最强的信号通路。见图5。
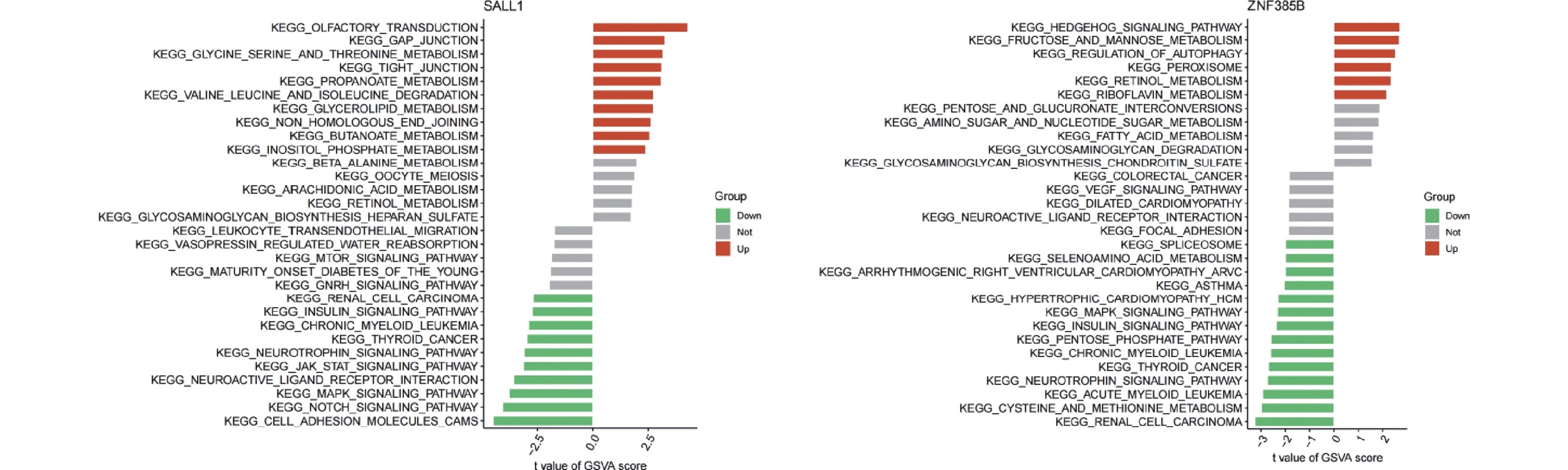
红色代表该基因集在特征基因(SALL1或ZNF385B)高表达分组中上调,绿色代表该基因集下降。
Red indicates that the gene set is up-regulated in the subgroup with high expression of the feature gene (SALL1 or ZNF385B), while green indicates that the gene set is down-regulated.
2.6 免疫浸润分析
应用quanTIseq算法预测样本的免疫浸润状况,探究关键特征基因与不同免疫细胞的关联情况。分析结果显示肿瘤样本中的M1型巨噬细胞和自然杀伤细胞(natural killer cell, NK)细胞含量比正常样本高,差异具有统计学意义。这提示RB样本中肿瘤微环境中的免疫反应较为活跃,这种反应有助于抑制肿瘤生长。进一步的分析显示12个关键特征基因与免疫细胞存在密切关联。见图6。
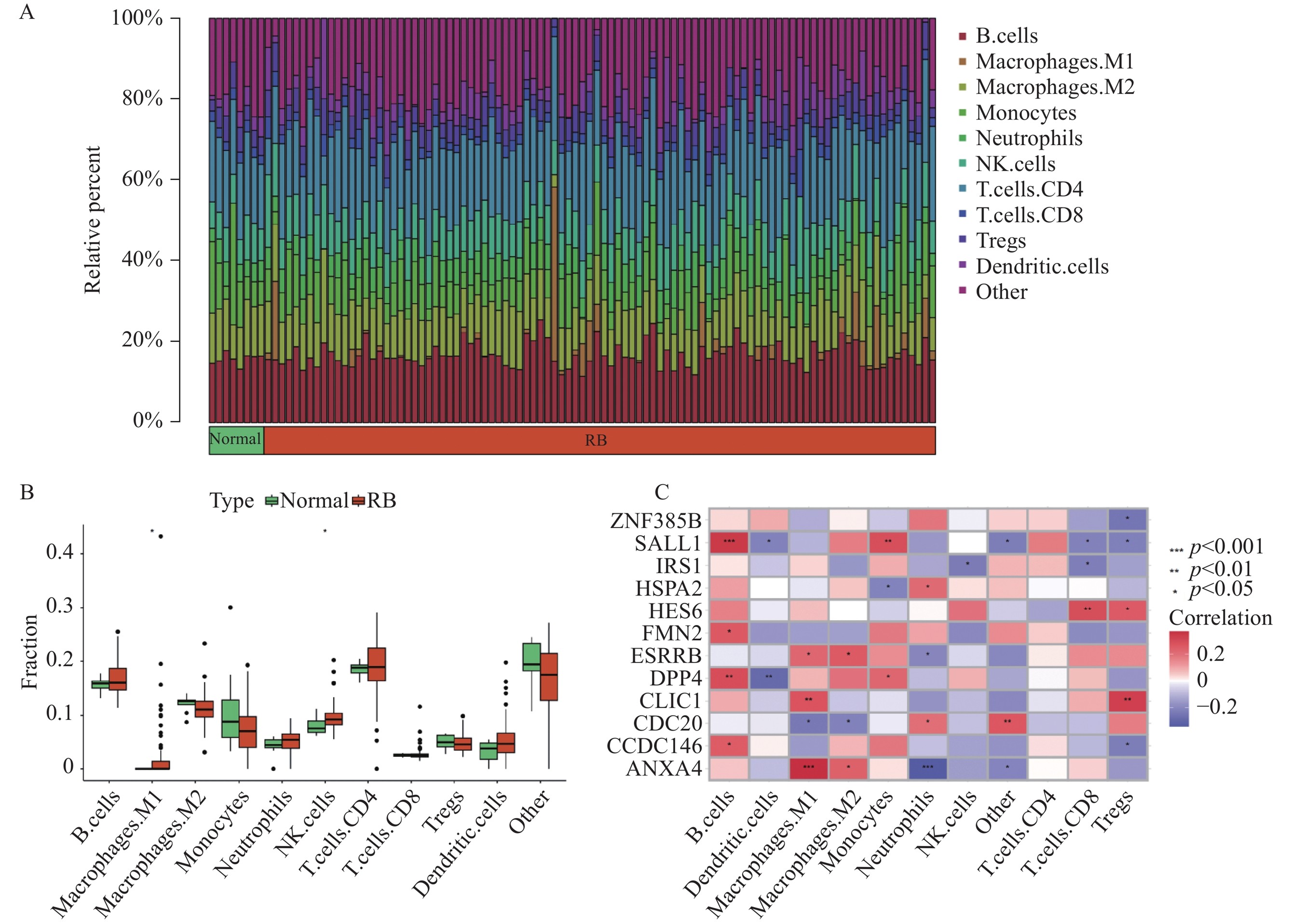
(A)不同样本中的免疫细胞含量条形图;(B)正常样本和肿瘤样本的免疫细胞含量差异箱式图;(C)12个关键特征基因与免疫细胞的关联图。
(A) Bar plot illustrating the relative proportions of various immune cell types across different samples. (B) Box plots comparing the infiltration levels of immune cells between normal and tumor samples. (C) Correlation heatmap between the 12 key feature genes and immune cell abundances.
2.7 单细胞数据分析
应用TISCH2平台,RB的GSE166173单细胞RNA测序数据集被纳入研究。分析显示Müller胶质细胞和视网膜细胞的占比最高。进一步分析展示了12个关键特征基因在不同细胞中正常样本和肿瘤样本的表达差异。见图7。
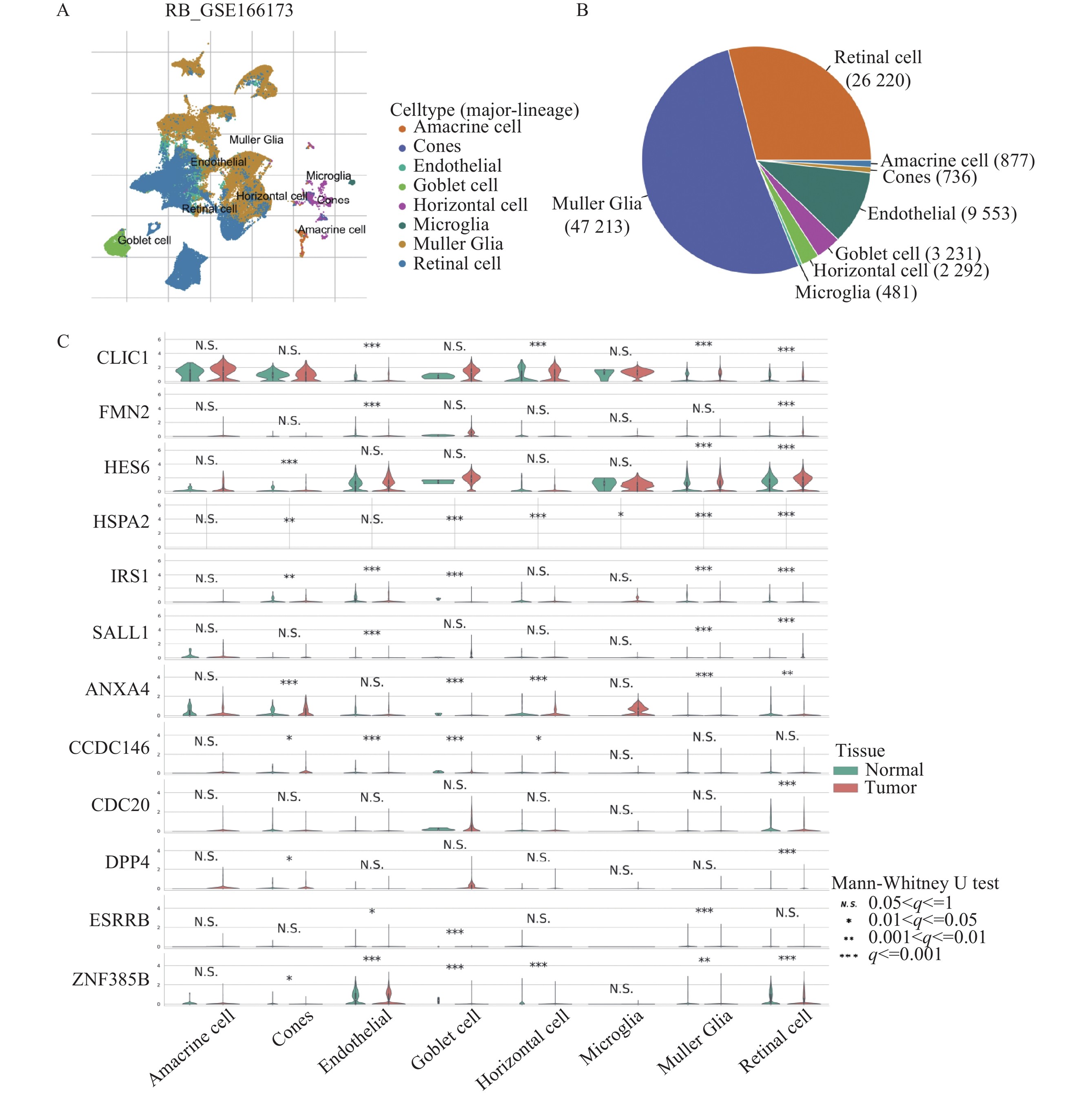
(A) UMAP图展示不同细胞亚型的分布情况;(B)饼图揭示不同细胞亚型的占比;(C) 12个关键特征基因在不同细胞亚型中肿瘤样本与正常样本的表达差异。
(A) UMAP projection colored by distinct cell subtypes. (B) Pie chart displaying the proportional abundance of each cell subtype. (C) Violin plots comparing the expression levels of the 12 key feature genes between tumor and normal samples across different cell subtypes.
2.8 药物富集分析
基于12个RB的关键特征基因,应用DSigDB数据库进行了治疗药物/化合物预测。分析结果显示排名靠前的4个药物:格列本脲、环磷酰胺、硫唑嘌呤和大豆苷元。见图8。这部分的分析预测了潜在的RB敏感治疗药物,为后续的药物研究提供了有价值的依据。
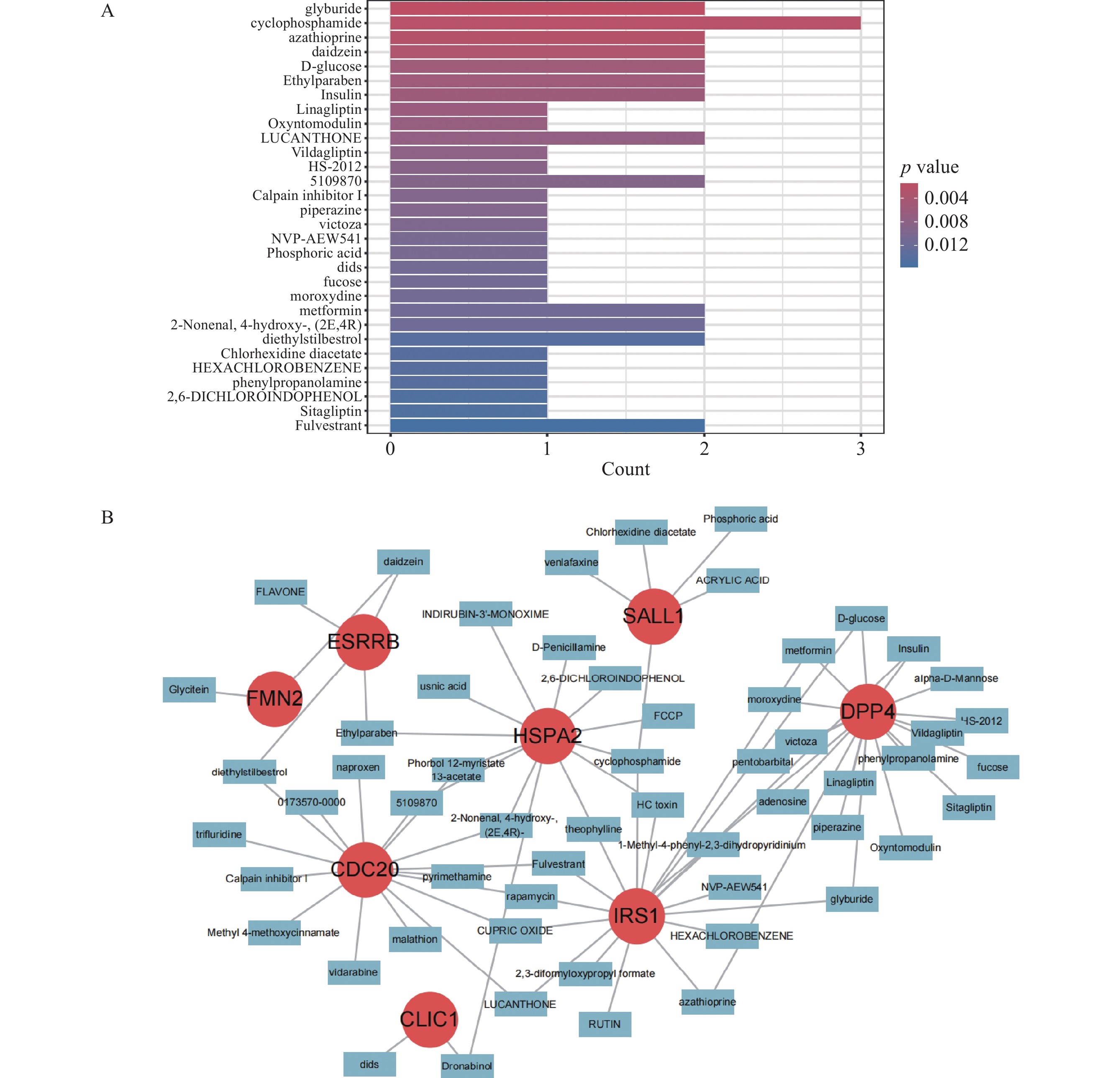
(A)药物富集分析条形图;(B)关键特征基因-敏感药物网络图。
(A) Bar plot of the enriched drugs. (B) Network of the key feature genes and their correlated sensitive drugs.
3 讨论
RB是一种起源于光感受器前体细胞的恶性肿瘤,在新生儿中的发病率约为1/17 000,每年预计新增约8 000例[12]。RB的发生、发展有着独特的遗传景观,临床实践证明基于RB1基因等分子的疾病分型对于RB的诊治具有重要的指导意义,因此基于现代多组学技术进一步挖掘RB疾病中的关键基因对细化疾病分型、深入了解分子机制和开发新治疗药物具有重要作用。
本研究纳入了3个来源于GEO的RB微阵列芯片数据集,通过去除批次差异合并后形成一个含有104个样本的研究队列,通过R语言软件完成mRNA的一系列分析。基因差异分析显示RB样本和正常视网膜样本间存在556个差异基因,说明肿瘤组织中存在明显的异常表达谱图。进一步的KEGG通路富集分析显示,这些基因参与了p53信号通路和细胞衰老等生物过程。GO基因功能分析显示这些基因主要与微管蛋白结合和染色体分离等生物过程有关。越来越多的证据表明p53信号通路与RB的发生、发展密切相关。许多基因处于p53-p21-pRb信号通路下,DNA复制和修复过程与之密切相关,其中p21在调节pRb磷酸化中起到关键作用[13-14]。
紧接着,本研究结合WGCNA和SVM方法来筛选关键的特征基因。WGCNA分析是一种系统生物学方法,用于描述微阵列样本中基因之间的关联模式。这方法可用于查找高度相关基因的模块,使用模块特征基因与外部样本特征相关联,应用于识别候选生物标志物或治疗靶点[9]。在本研究中WGCNA分析的软阈值(soft thresholding)为4,经过计算得到了1个核心的基因模块,其中turquoise模块与RB呈正相关,初步得到了853个特征基因。此外,本研究应用了SVM-RFE算法得到了16个特征基因,SVM可输出各基因对分类超平面的贡献度,间接反映基因重要性。通过迭代剔除对分类贡献最小的基因,保留关键特征基因。将该2种机器学习方法取交集得到12个关键特征基因。在单细胞水平分析中,关键特征基因主要在Müller胶质细胞和视网膜细胞亚型中表现出表达差异。在这当中SALL 1和ZNF385B分别是显著上调和显著下调的特征基因。SALL 蛋白是一个由4种保守的C2H2 锌指转录因子组成的家族,在胚胎发育过程中的器官形成中起关键作用。它们调节细胞增殖、存活、迁移等。SALL1是调节干细胞多能性的转录网络的成员,据文献报道,过表达SALL1基因在不同的癌症中表现出不同的效应。在乳腺癌细胞中过表达SALL1会抑制肿瘤细胞生长和增殖[15]。而在急性髓性白血病中,过表达的SALL1与不良预后有关[16]。目前,尚无SALL1过表达与RB的预后相关的研究报道。ZNF385B同样是锌指蛋白家族的一员,可能影响机体免疫系统、肿瘤细胞存活并改变肿瘤微环境,进而改变肿瘤的发生、发展。而ZNF385B基因在RB中的调控作用尚未见报道。本研究分析得出RB肿瘤样本中过表达的SALL1与甘氨酸、丝氨酸和苏氨酸代谢等通路上调存在关联。氨基酸代谢异常是癌症的重要特征之一,肿瘤细胞通过重编程代谢途径来支持其快速增殖、侵袭和免疫逃逸。ZNF385B基因的低表达会上调肾细胞癌和急性髓性白血病等疾病的相关通路。因此,这些关键特征基因可能是潜在的新靶点,在今后的研究中需要进一步结合体内外生物学实验进行验证。
研究发现,TME是肿瘤细胞、免疫细胞、内皮细胞与成纤维细胞等不同细胞类型之间复杂相互作用的结果,可调控多种肿瘤的发生和转移[17]。因此,了解肿瘤内异质性和微环境与肿瘤发生及恶性程度的关系具有重要意义[18-19]。研究表明,凋亡和炎症途径中的免疫失调和异常在RB肿瘤发展中发挥重要作用[20-21]。免疫疗法的发展有助于在不影响视力的情况下根除肿瘤,这将在很大程度上改善RB患者的生活质量[22]。基于此,本研究计算了RB肿瘤样本中的免疫浸润状况以分析免疫微环境的变化,探究不同免疫细胞的含量与疾病的关联。分析表明RB肿瘤样本中含量最高的是巨噬细胞、NK细胞和T细胞。肿瘤微环境中以高含量的巨噬细胞浸润为主要特征,Wu等[23]基于单细胞测序表明RB中TME主要由肿瘤相关巨噬细胞(Tumor-Associated Macrophages, TAMs)、星形胶质细胞样细胞和CAF细胞组成。巨噬细胞在组织稳态、组织重塑、免疫反应和癌症进展中起重要作用。在此研究肿瘤样本中的M1 型巨噬细胞和NK细胞高于正常样本,这提示肿瘤组织中存在强烈的抗肿瘤免疫反应。M1 型是促炎巨噬细胞,通过促进强大的 T 和NK细胞抗肿瘤反应来启动免疫反应、破坏组织完整性并抑制肿瘤进展[24]。促炎性M1型巨噬细胞释放大量促炎细胞因子,包括白细胞介素(interleukin, IL)-1b、IL-6、IL-12、IL-23、IFN-β和TNF-ɑ,这些因子同时驱动Th1免疫反应[25]。高度活化的M1型巨噬细胞也与NK细胞存在密切关联。M1型巨噬细胞释放的白细胞IL-1β、IFN-β和IL-23会促进NK细胞p44相关蛋白(NKp44)及NKG2D的表达,进而对靶细胞发动细胞毒性反应,发挥抗肿瘤效应[26-27]。M1型巨噬细胞和NK细胞的交互作用共同构成了抗肿瘤防线。此外,本研究分析了关键差异基因和免疫细胞间的关联。在敏感药物预测中,本研究发现格列本脲、环磷酰胺、硫唑嘌呤和大豆苷元可能是潜在的敏感药物。本研究的局限在于缺少基础实验验证,在后续的研究中将采用具体的实验来验证关键差异基因的表达情况和具体作用机制。
综上所述,本研究基于生物信息学的方法应用GEO数据集鉴定了RB中的关键差异基因,进一步分析了肿瘤样本中的免疫浸润情况并预测了敏感药物,为进一步了解该疾病的分子机制和开发新型药物提供了有价值的参考依据。
声明
本稿件在研究和论文撰写中未使用生成式人工智能,所有作者对内容的真实性、完整性和科学性负责。所有科学贡献和智力劳动均由所有作者共同完成。
利益冲突
所有作者均声明不存在利益冲突。
开放获取声明
本文适用于知识共享许可协议(Creative Commons),允许第三方用户按照署名(BY)-非商业性使用(NC)-禁止演绎(ND)(CC BY-NC-ND)的方式共享,即允许第三方对本刊发表的文章进行复制、发行、展览、表演、放映、广播或通过信息网络向公众传播,但在这些过程中必须保留作者署名、仅限于非商业性目的、不得进行演绎创作。
基金
参考文献
1. Liu J, Ottaviani D, Sefta M, et al. A high-risk retinoblastoma subtype with stemness features, dedifferentiated cone states and neuronal/ganglion cell gene expression[J]. Nat Commun, 2021, 12(1): 5578. DOI: 10.1038/s41467-021-25792-0.
2. Broaddus E, Topham A, Singh AD. Incidence of retinoblastoma in the USA: 1975-2004[J]. Br J Ophthalmol, 2009, 93(1): 21-23. DOI: 10.1136/bjo.2008.138750.
3. Cruz-Gálvez CC, Ordaz-Favila JC, Villar-Calvo VM, et al. Retinoblastoma: review and new insights[J]. Front Oncol, 2022, 12: 963780. DOI: 10.3389/fonc.2022.963780.
4. Schaiquevich P, Francis JH, Cancela MB, et al. Treatment of retinoblastoma: what is the latest and what is the future[J]. Front Oncol, 2022, 12: 822330. DOI: 10.3389/fonc.2022.822330.
5. Fabian ID, Abdallah E, Abdullahi SU, et al. Global retinoblastoma presentation and analysis by national income level[J]. JAMA Oncol, 2020, 6(5): 685-695. DOI: 10.1001/jamaoncol.2019.6716.
6. Munier FL, Beck-Popovic M, Chantada GL, et al. Conservative management of retinoblastoma: Challenging orthodoxy without compromising the state of metastatic grace. “Alive, with good vision and no comorbidity”[J]. Prog Retin Eye Res, 2019, 73: 100764. DOI: 10.1016/j.preteyeres.2019.05.005.
7. Saunders NA, Simpson F, Thompson EW, et al. Role of intratumoural heterogeneity in cancer drug resistance: molecular and clinical perspectives[J]. EMBO Mol Med, 2012, 4(8): 675-684. DOI: 10.1002/emmm.201101131.
8. Dagogo-Jack I, Shaw AT. Tumour heterogeneity and resistance to cancer therapies[J]. Nat Rev Clin Oncol, 2018, 15(2): 81-94. DOI: 10.1038/nrclinonc.2017.166.
9. Langfelder P, Horvath S. WGCNA: an R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9: 559. DOI: 10.1186/1471-2105-9-559.
10. Han Y, Wang Y, Dong X, et al. TISCH2: expanded datasets and new tools for single-cell transcriptome analyses of the tumor microenvironment[J]. Nucleic Acids Res, 2023, 51(D1): D1425-D1431. DOI: 10.1093/nar/gkac959.
11. Yoo M, Shin J, Kim J, et al. DSigDB: drug signatures database for gene set analysis[J]. Bioinformatics, 2015, 31(18): 3069-3071. DOI: 10.1093/bioinformatics/btv313.
12. Aschero R, Simao M, Catala-Mora J, et al. Risk factors for extraocular relapse in retinoblastoma[J]. Semin Ophthalmol, 2025, 40(6): 474-484. DOI: 10.1080/08820538.2025.2450682.
13. Engeland K. Cell cycle regulation: p53-p21-RB signaling[J]. Cell Death Differ, 2022, 29(5): 946-960. DOI: 10.1038/s41418-022-00988-z.
14. McEvoy JD, Dyer MA. Genetic and epigenetic discoveries in human retinoblastoma[J]. Crit Rev Oncog, 2015, 20(3-4): 217-225. DOI: 10.1615/critrevoncog.2015013711.
15. Ma C, Wang F, Han B, et al. SALL1 functions as a tumor suppressor in breast cancer by regulating cancer cell senescence and metastasis through the NuRD complex[J]. Mol Cancer, 2018, 17(1): 78. DOI: 10.1186/s12943-018-0824-y.
16. Salman H, Shuai X, Nguyen-Lefebvre AT, et al. SALL1 expression in acute myeloid leukemia[J]. Oncotarget, 2017, 9(7): 7442-7452. DOI: 10.18632/oncotarget.23448.
17. Yuan Y, Jiang YC, Sun CK, et al. Role of the tumor microenvironment in tumor progression and the clinical applications (Review)[J]. Oncol Rep, 2016, 35(5): 2499-2515. DOI: 10.3892/or.2016.4660.
18. Collin J, Queen R, Zerti D, et al. Dissecting the transcriptional and chromatin accessibility heterogeneity of proliferating cone precursors in human retinoblastoma tumors by single cell sequencing-opening pathways to new therapeutic strategies?[J]. Invest Ophthalmol Vis Sci, 2021, 62(6): 18. DOI: 10.1167/iovs.62.6.18.
19. Yang J, Li Y, Han Y, et al. Single-cell transcriptome profiling reveals intratumoural heterogeneity and malignant progression in retinoblastoma[J]. Cell Death Dis, 2021, 12(12): 1100. DOI: 10.1038/s41419-021-04390-4.
20. Kulbay M, Tuli N, Mazza M, et al. Oncolytic viruses and immunotherapy for the treatment of uveal melanoma and retinoblastoma: the current landscape and novel advances[J]. Biomedicines, 2025, 13(1): 108. DOI: 10.3390/biomedicines13010108.
21. Kaewkhaw R, Rojanaporn D. Retinoblastoma: etiology, modeling, and treatment[J]. Cancers, 2020, 12(8): 2304. DOI: 10.3390/cancers12082304.
22. Mao P, Shen Y, Xu X, et al. Comprehensive analysis of the immune cell infiltration landscape and immune-related methylation in retinoblastoma[J]. Front Genet, 2022, 13: 864473. DOI: 10.3389/fgene.2022.864473.
23. Wu C, Yang J, Xiao W, et al. Single-cell characterization of malignant phenotypes and microenvironment alteration in retinoblastoma[J]. Cell Death Dis, 2022, 13(5): 438. DOI: 10.1038/s41419-022-04904-8.
24. Kerneur C, Cano CE, Olive D. Major pathways involved in macrophage polarization in cancer[J]. Front Immunol, 2022, 13: 1026954. DOI: 10.3389/fimmu.2022.1026954.
25. Ostuni R, Kratochvill F, Murray PJ, et al. Macrophages and cancer: from mechanisms to therapeutic implications[J]. Trends Immunol, 2015, 36(4): 229-239. DOI: 10.1016/j.it.2015.02.004.
26. Mattiola I, Pesant M, Tentorio PF, et al. Priming of human resting NK cells by autologous M1 macrophages via the engagement of IL-1β, IFN-β, and IL-15 pathways[J]. J Immunol, 2015, 195(6): 2818-2828. DOI: 10.4049/jimmunol.1500325.
27. Melaiu O, Lucarini V, Cifaldi L, et al. Influence of the tumor microenvironment on NK cell function in solid tumors[J]. Front Immunol, 2020, 10: 3038. DOI: 10.3389/fimmu.2019.03038.